Physical Address
304 North Cardinal St.
Dorchester Center, MA 02124
Physical Address
304 North Cardinal St.
Dorchester Center, MA 02124
In questo articolo vedremo come realizzare istogrammi e density plot in R.
È possibile creare istogrammi con la funzione hist(x) dove x è un vettore numerico di valori da visualizzare.
hist(x, main="Titolo del grafico", ylab="label asse y", xlab="label asse x")
Di default, hist usa le frequenze, non la densità, sull’asse delle y. Per ottenere un vero e proprio istogramma passeremo alla funzione l’argomento freq, specificando che non le vogliamo.
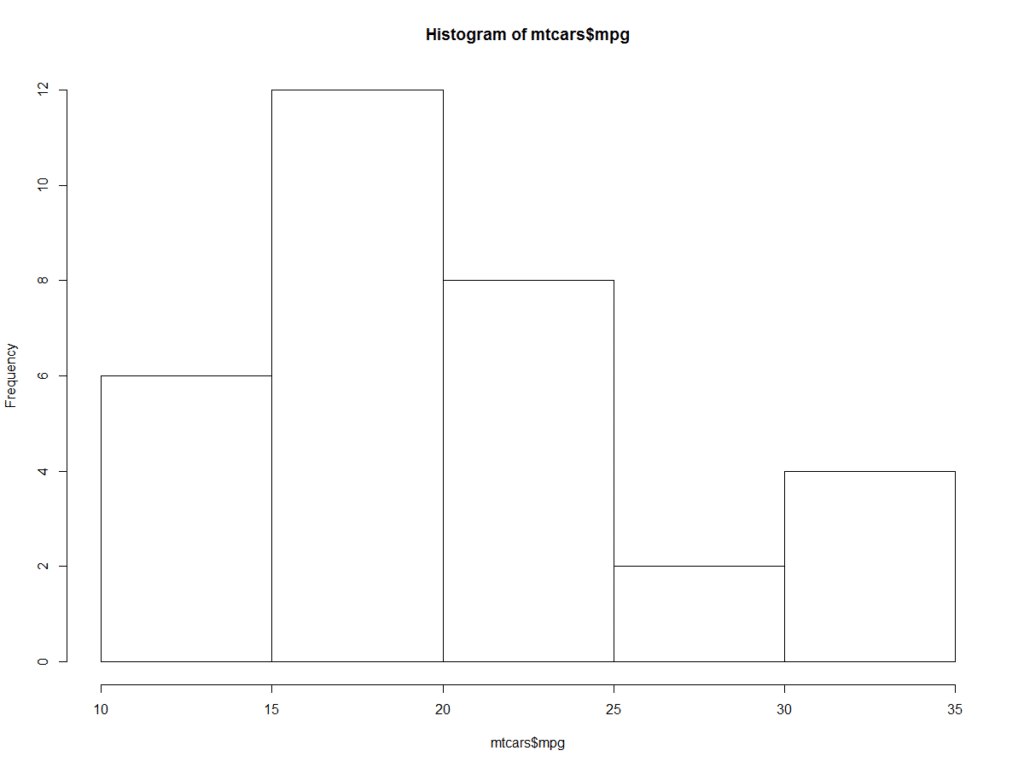
# Esempio Istogramma
hist(mtcars$mpg)
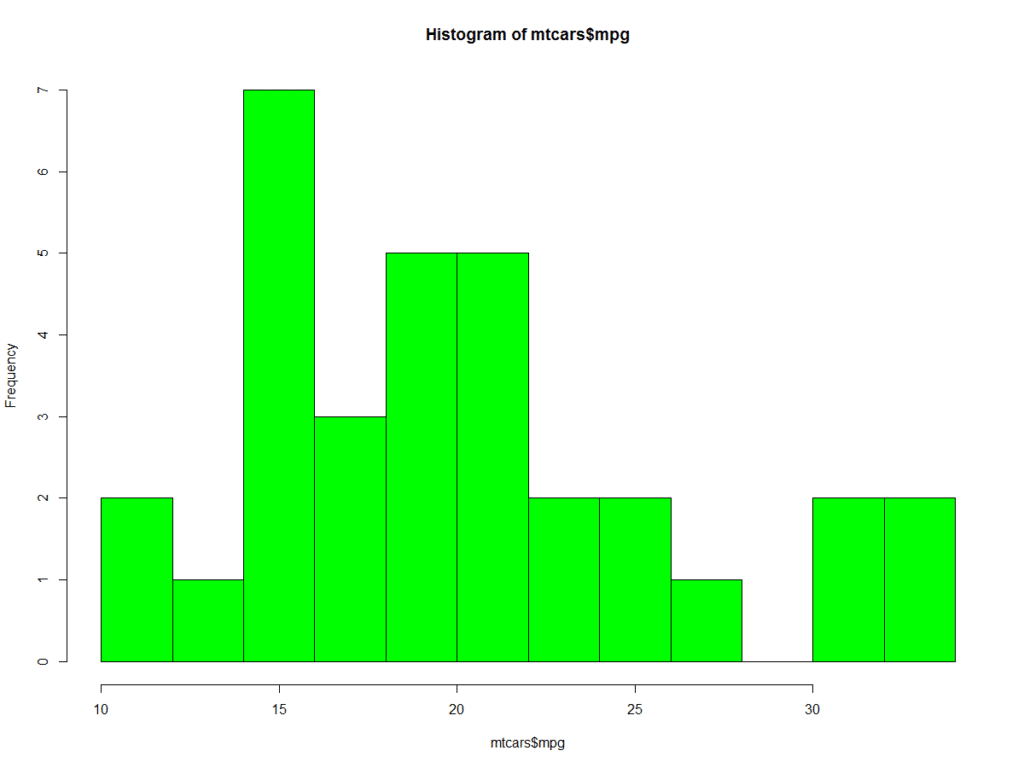
#Istogramma colorato con diverso numero di contenitori
hist(mtcars$mpg, breaks=9, col="green")
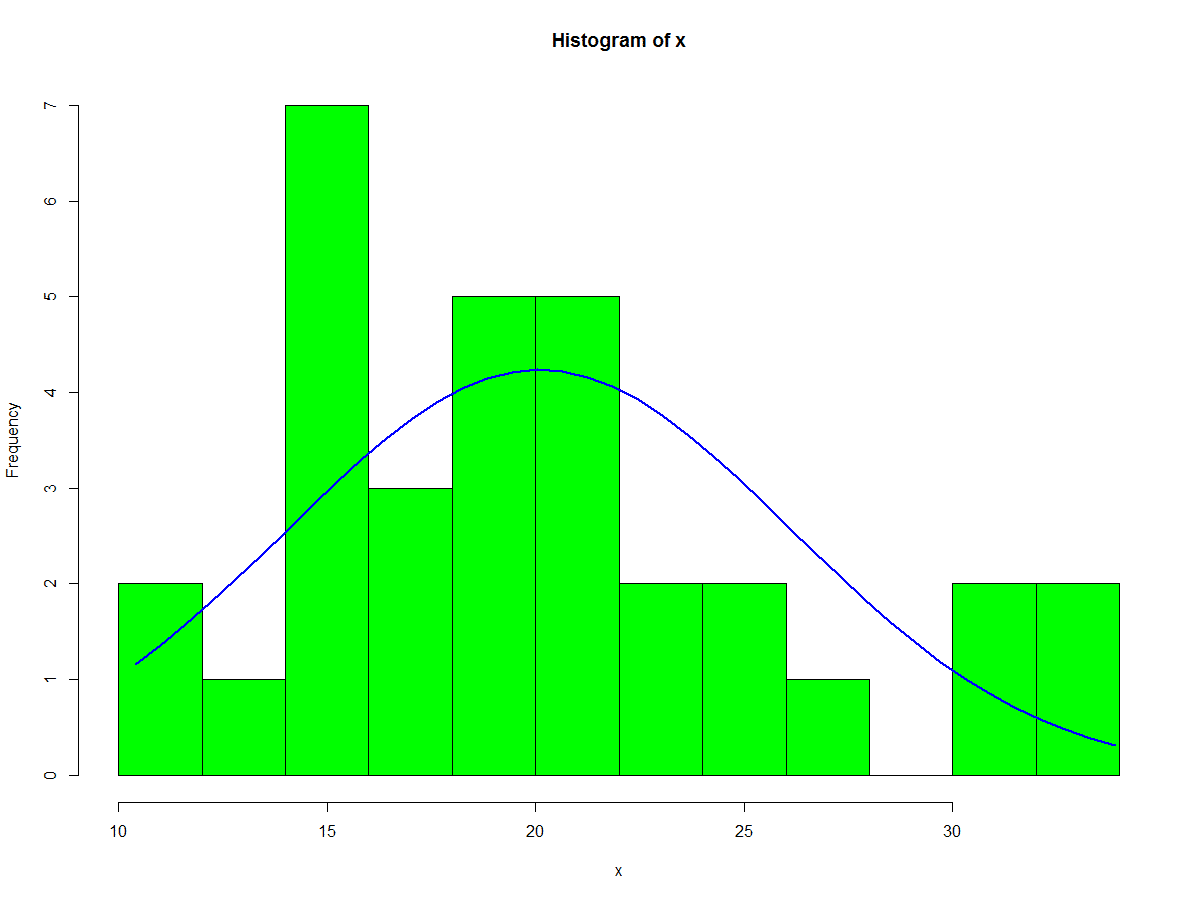
# Aggiungiamo una curva normale
x <- mtcars$mpg
h<-hist(x, breaks=9, col="green")
xfit<-seq(min(x),max(x),length=40)
yfit<-dnorm(xfit,mean=mean(x),sd=sd(x))
yfit <- yfit*diff(h$mids[1:2])*length(x)
lines(xfit, yfit, col="blue", lwd=2)
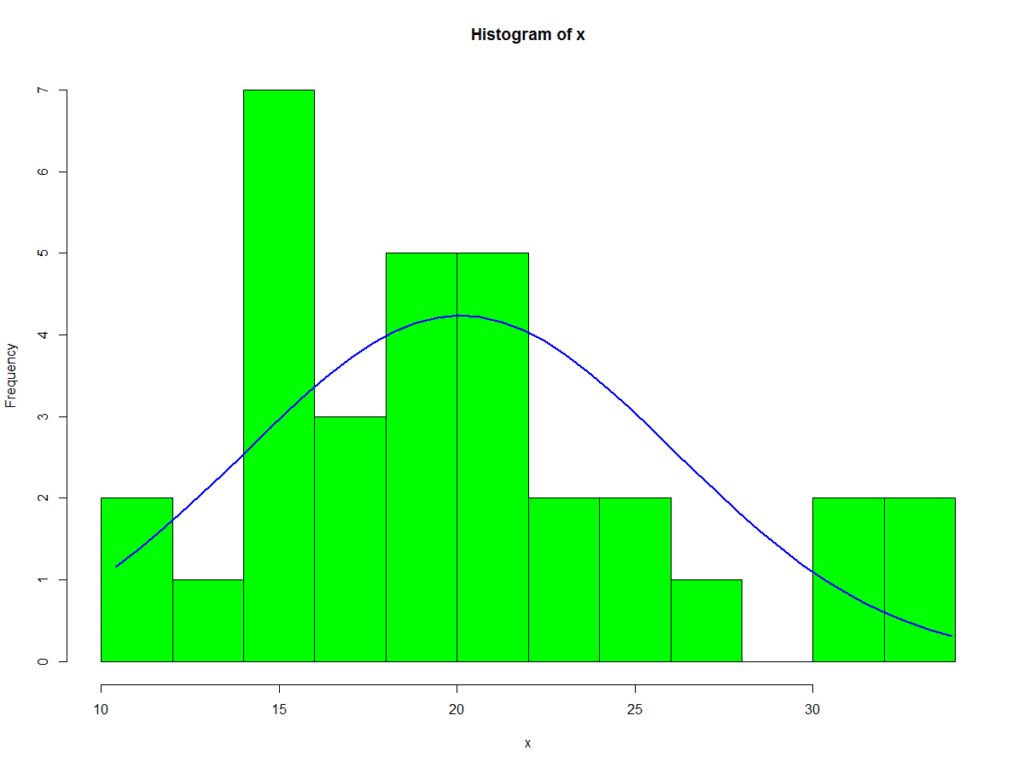
Gli istogrammi, comunque, non sono un ottimo metodo per determinare la forma di una distribuzione perché è molto influenzata dal numero di contenitori usati.
Altri argomenti della funzione plot
nclass=n: genera un isotgramma con un numero n di classi
probability=TRUE le colonne rappresentano frequenze relative invece che assolute.
hist(x, …)
# S3 method for default
hist(x, breaks = "Sturges",
freq = NULL, probability = !freq,
include.lowest = TRUE, right = TRUE,
density = NULL, angle = 45, col = NULL, border = NULL,
main = paste("Histogram of" , xname),
xlim = range(breaks), ylim = NULL,
xlab = xname, ylab,
axes = TRUE, plot = TRUE, labels = FALSE,
nclass = NULL, warn.unused = TRUE, …)
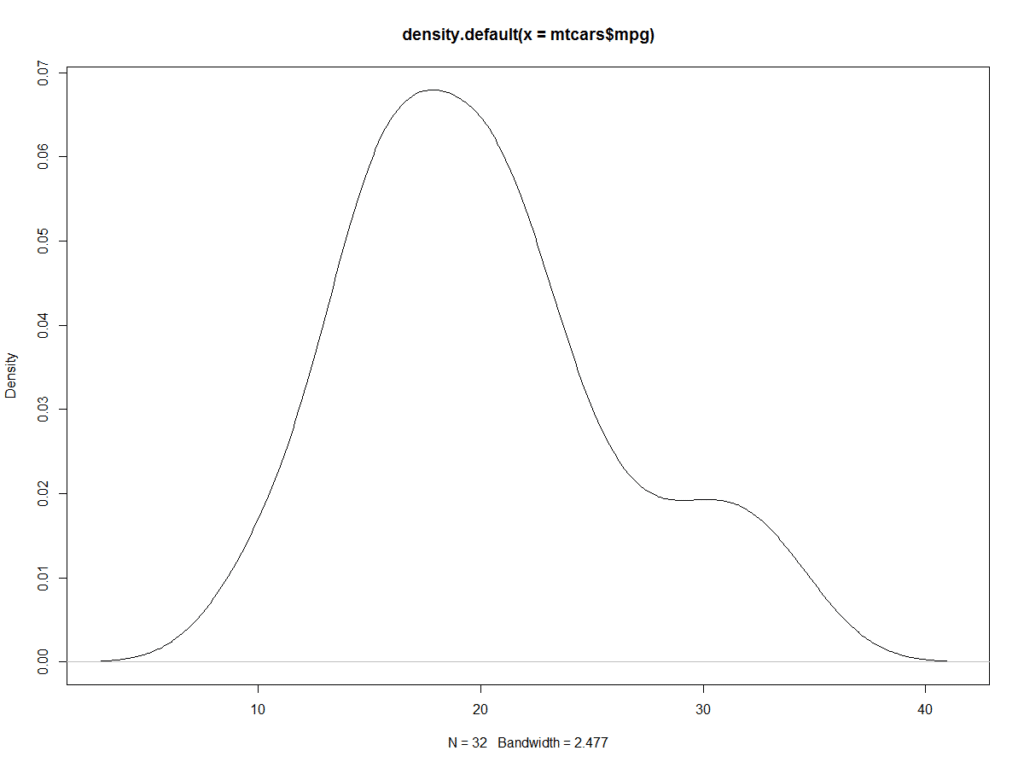
I Kernel denity plot in R rappresentano un modo molto efficace per visualizzare la distribuzione di una variabile. Basta visualizzare il grafico della density(x) dove x è un vettore numerico.
density(x, …)
# S3 method for default
density(x, bw = "nrd0", adjust = 1,
kernel = c("gaussian", "epanechnikov", "rectangular",
"triangular", "biweight",
"cosine", "optcosine"),
weights = NULL, window = kernel, width,
give.Rkern = FALSE,
n = 512, from, to, cut = 3, na.rm = FALSE, …)
# Kernel density plot
d <- density(mtcars$mpg)
plot(d)
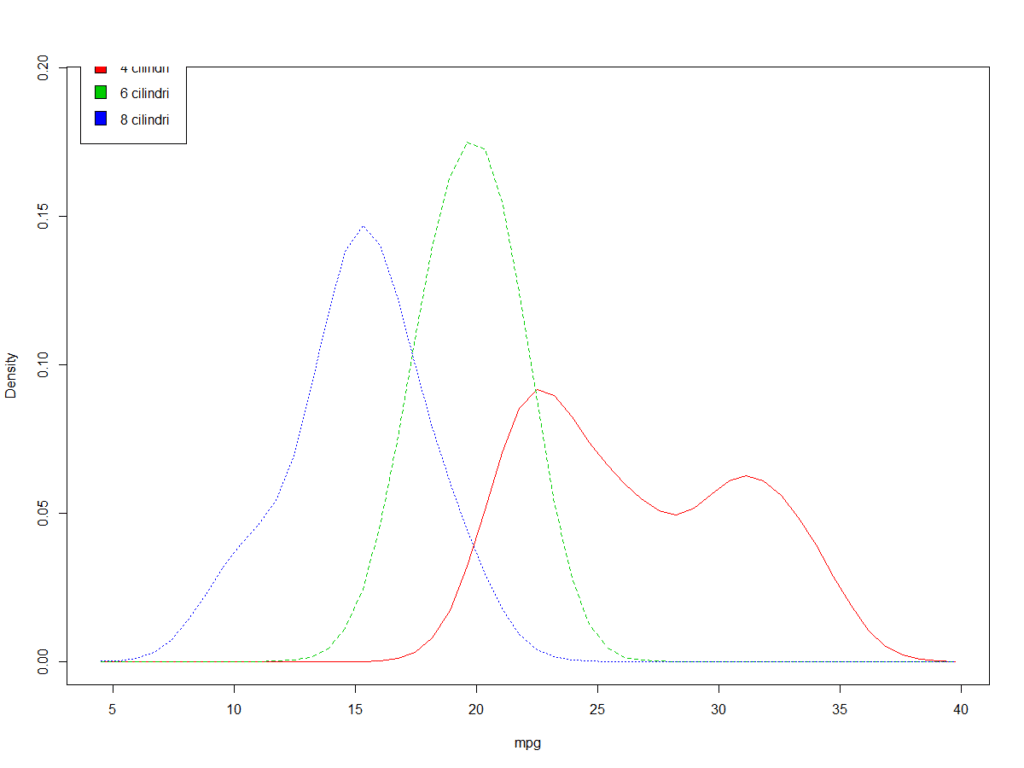
Attraverso la funzione sm.density.compare() nel package sm è possibile sovrapporre i grafici di densità kernel di due o più gruppi.
Il formato è sm.density.compare (x, factor) dove x è un vettore numerico e factor è la variabile di raggruppamento.
library(sm)
attach(mtcars)
cyl.f <- factor(cyl, levels= c(4,6,8),
labels = c("4 cilindri", "6 cilindri", "8 cilindri"))
sm.density.compare(mpg, cyl )
colfill<-c(2:(2+length(levels(cyl.f))))
legend(locator(1), levels(cyl.f), fill=colfill)
Guarda tutti i tutorial sui Grafici oppure torna su R tutorial.